Ça s’est passé au cours d’une session de formation d’une durée de deux semaines conduite par le Pr. Christian Happi, Directeur du Centre d’Excellence Africain pour la Génomique des Maladies Infectieuses (ACEGID), et son équipe.
Avec la pandémie de COVID-19, le séquençage de nouvelle génération (NGS) est devenu une technique indispensable en génétique et en génomique. C’est dans ce contexte qu’intervient cette session de renforcement des capacités qui a eu lieu du 18 Février au 02 Mars 2022, au CRID, à Yaoundé, la capitale politique du Cameroun. Les travaux ont duré quatorze jours au total. Ils étaient placés sous la coordination des experts de l’ACEGID de la « Redeemer’s University » d’Ede au Nigeria. Une conférence sur le Séquençage de Nouvelle Génération (NGS) et la batterie d’opportunités y afférentes ont été données par le professeur Christian Happi à l’entame de cette session de formation. Au cours de celle-ci, 6 chercheurs du CRID ont été initiés à la mise en place des plateformes de séquençage Illumina Miseq et Nanopore MinION destinées à séquencer le SARS-CoV-2 et les arbovirus.
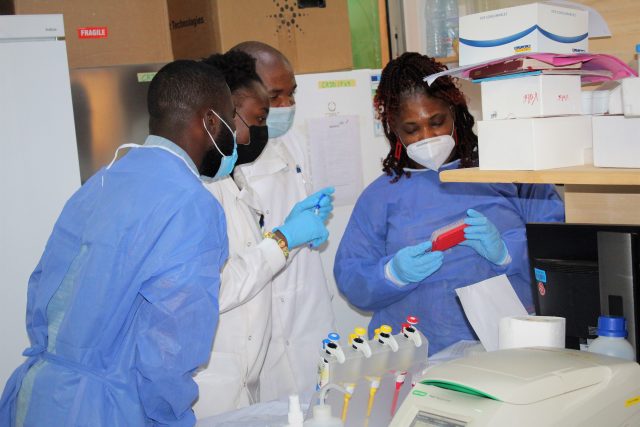
Session de pratique du séquençage de nouvelle génération
13 jours de travaux pratiques
Ce programme de renforcement des capacités a integré aussi bien des enseignements théoriques que des travaux pratiques relatifs aux technologies NGS actuellement disponibles au CRID. Parmi celles-ci on peut citer les plateformes de séquençage à lecture courte illumina Miseq et Oxford Nanopore MinION à lecture longue. Les techniciens de laboratoire et des chercheurs postdoctoraux ont constitué l’essentiel des personnels formés à la manipulation de ces machines. Le but ici étant d’approfondir la compréhension du séquençage à haut débit et de permettre au CRID d’effectuer le séquençage sur site. En somme, cette formation a intégré des présentations, des discussions et des sessions pratiques couvrant la préparation de la base de données, du séquençage et de l’analyse des données. Concrètement, la session pratique impliquait le séquençage d’échantillons de COVID-19 précédemment collectés à l’aide du kit COVIDseq illumina sur le Miseq et du protocole ONT minuit SARS-COV-2 sur le MinION. Des aspects essentiels tels que la création des bases de données de haute qualité à partir d’échantillons, la préparation des cellules d’écoulement pour le séquençage, l’effectivité des cycles de séquençage sur les différentes plates-formes et l’identification des indicateurs du bon fonctionnement d’un cycle ont été abordés au détail près. Des outils open-source tels qu’ IDseq, pangolin COVID-19, EDGE bioinformatics et Nextclade ont été utilisés dans le cadre de l’analyse des données. Un examen métagénomique 16s a également été réalisé sur des moustiques pour vérifier leur diversité microbienne.

Moment de la leçon inaugurale du Prof Christian Happy
Une session de formation bien réussie
Le succès de cette formation s’est traduit par la génération de données de métagénomique 16s et de certains génomes COVID; engendrés par les plateformes de séquençage. Au sortir de cet atelier, d’excellents retours ont été enregistrés aussi bien chez les formateurs de l’ACEGID, que chez les chercheurs du CRID. Ce qui donne de considérer que la collaboration entre ces deux partenaires à la recherche à de beaux jours devant elle. En effet, le CRID et l’ACEGID entendent renforcer leurs échanges afin de faire des progrès encore plus importants dans la lutte contre les maladies infectieuses. Il faut relever que la tenue de cet atelier est la concrétisation d’une de ces missions sacrées reconnues au CRID; celle de mener des recherches de haute qualité et internationalement approuvées dans le secteur des maladies au Cameroun et en Afrique, de contribuer à la formation et au renforcement des savoir-faire de la nouvelle génération de scientifiques africains.